Как известно, для того чтобы закодировать огромный объем информации в генетическом коде, используется всего 4 нуклеиновых кислоты: аденин, гуанин, тимин и цитозин. В генетическом коде они обозначаются соответствующими буквами — А, Г, Т и Ц. Таким образом, можно сказать, что «генетический алфавит» состоит из 4 букв, и до последнего времени считалось, что изменить его нельзя, однако группа ученых из Института Скриппса впервые сумела дополнить его двумя новыми буквами и при этом оставить его полностью функционирующим.
У всех живых организмов вышеописанные нуклеиновые кислоты соединяются друг с другом не абы как, а по принципу комплиментарности. То есть они как бы «смотрят» друг на друга, причем напротив А всегда должен стоять Т, а напротив Г – Ц, и никак иначе. Но это еще полдела. Эти буквы должны «складываться в слова», которые носят название триплеты – особые комбинации, благодаря которым и происходят все основные моменты вроде считывания информации, кодирования белков и так далее. Несколько лет назад журнал Science опубликовал статью, в которой описывался опыт, в ходе которого транспортные РНК приносили к ДНК новую аминокислоту, которая была распознана и встраивалась в код. При этом эта кислота была лишь одна, не имела пары и новую функцию не выполняла.
В новом же изыскании исследователи из компании Synthorx использовали два новых азотистых основания (обозначенные X и Y). Они в двуцепочечной молекуле ДНК расположены друг напротив друга, как и стандартные 4 основания, но, в отличие от них, «новые буквы» соединяются не водородными связями, а гидрофобными. Причем, встроив два новых основания в ДНК бактерий, последние смогли их воспроизводить, но такие бактерии поначалу делились медленнее обычного и иногда заменяли новую ДНК «традиционной». Сейчас уже выведены бактерии, которые без проблем воспроизводят новую ДНК. Осталось только придумать для этих букв новые триплеты.
«Если посчитать, сколько комбинаций-триплетов можно получить, имея на руках четыре буквы, то мы получим 64 комбинации, причем, добавив всего лишь две буквы, мы расширяем число возможных генетических слов до 216, а в результате появится возможность кодировать еще 172 аминокислоты, что открывает просто бескрайний простор для биоинженерии».
С двумя буквами подопытные бактерии получили и два новых генетических слова.
У всех живых организмов, от бактерий до человека, генетический алфавит состоит из четырех молекулярных букв – А, Т, Г, Ц. Буквы эти – азотистые основания аденин, тимин, гуанин и цитозин. В двуцепочечной молекуле ДНК они пришиты к длиннейшим сахарофософатным «перилам» и смотрят друг на друга – то есть в пространство между цепочек, причем напротив А всегда должен стоять Т, а напротив Г – Ц.
Но сами по себе буквы ничего не значат – они должны складываться в слова. Слова в генетическом коде земной жизни состоят из трех букв, образующих между собой разные комбинации. Такие слова-триплеты соответствуют аминокислотам, и когда идет синтез белка, то специальные машины читают код тройками, и в соответствии с последовательностью троек строят последовательность аминокислот. (Там есть особенности с передачей информации от ДНК через матричную РНК к белоксинтезирующим машинам, но эти подробности мы сейчас опустим.)
Если посчитать, сколько комбинаций-триплетов можно получить, имея на руках четыре буквы, то мы получим 64 комбинации. Между тем в синтезе белков участвуют всего 20 аминокислот. То есть код сильно избыточен, и потому одной аминокислоте часто соответствуют два, три и больше генетических слов.
Рано или поздно биологам должны была прийти в голову мысль, что генетический код можно отредактировать на фундаментальном уровне, то есть приписать каким-то словам новые значения, а то и вовсе расширить алфавит и словарь. Смена значений слов кажется более сложной задачей, ведь тут нужно сначала лишить триплет прежнего значения (о том, как это делают, мы как-то писали), а уж потом приписать ему новое.
Тем не менее, несколько лет назад в Science вышла статья, в которой описывалось, как у бактерий кишечной палочки удалось присвоить одному из избыточных триплетов значение абсолютно новой аминокислоты. Поскольку аминокислоты к белоксинтезирующей машине подвозят специальные транспортные РНК (тРНК), то пришлось создать и новую тРНК, которая носила бы с собой новую аминокислоту и распознавала бы ее код во время синтеза полипептидной цепи.
Но тРНК не сами хватают свои аминокислоты, для этого есть специальные ферменты – аминоацил-тРНК-синтетазы, которые узнают только свои аминокислоты и соответствующие им тРНК. Понятно, что авторам работы пришлось создать тут и новый фермент, который бы соединял новую аминокислоту с новой тРНК.
Но можно, как мы сказали, начать редактировать код еще раньше – с алфавита. Иными словами, что мы сначала должны создать новую букву, то есть новое азотистое основание. На самом деле, с химической точки зрения тут особо сложного ничего нет, и создать новую букву можно, просто модифицировав какую-нибудь старую. (Более того, клетки сами модифицируют «буквы», и порой довольно сильно, правда, код от этого не меняется.)
Но мало синтезировать новую «букву», она должна нормально встроиться в нуклеиновую кислоту и участвовать во всех необходимых молекулярных процессах. Впервые такие «буквы» появились в конце 80-х годов прошлого века, то были модификации гуанина и цитозина; ДНК с ними нормально удваивалась и на ней нормально синтезировалась молекула-посредник – матричная РНК, которая несет информацию о белке к белоксинтезирующему аппарату.
Флойд Ромесберг (Floyd Romesberg) и его коллеги из Института Скриппса за последние двадцать лет создали несколько сотен новых букв, однако расширить с их помощью генетический код удалось только сейчас. В своей последней работе исследователи вместе с сотрудниками биотехнологической компании Synthorx использовали два новых азотистых основания, которые для простоты лучше назвать X и Y.
В двуцепочечной молекуле ДНК они стоят друг напротив друга, но при этом соединяются они не так, как все остальные: если А и Т, Г и Ц держатся друг за друга водородными связями, то X и Y удерживаются друг напротив друга гидрофобными силами, что добавляет им странности. Тем не менее, пара X и Y не выбивается из ряда прочих «традиционных» пар, а значит, не нарушает структуру ДНК – ее физические параметры (расстояния между цепями, размер шага спирали и т. д.) остаются по всей длине прежними, что очень важно для взаимодействия с белками и вообще для целостности молекулы.
Поначалу, впрочем, бактерии с такой ДНК делились медленнее обычного и норовили заменить «нетрадиционную» ДНК нормальной. Однако исследователи все-таки доработали новые буквы, так что они перестали доставлять неудобство бактериям. Теперь осталось только сделать из них новые слова-триплеты, которые кодировали бы две аминокислоты. В качестве новых аминокислот выбрали модификации лизина и фенилаланина. Новые триплеты генетических букв оказались такими: AXC (то есть аденин-X-цитозин) и GYT (гуанин-Y-тимин). Их внедрили в ДНК, кодирующую зеленый флуоресцентный белок, а ДНК отправили в бактериальную клетку – и клетка засветилась зеленым. При этом, разумеется, у бактерий были вышеупомянутые ферменты и тРНК, чтобы обслуживать новые триплеты в коде и новые аминокислоты, а сами аминокислоты нужно было добавлять в питательную среду. Полностью результаты описаны в статье в Nature.
Возникает вопрос, зачем вообще нужно столь фундаментально вмешиваться в генетический алфавит и словарь. Здесь все просто: с новыми аминокислотами можно создавать новые белковые молекулы, сконструированные для конкретных задач. Конечно, белки модифицировали в лабораториях и раньше, но, внедрившись в генетический код, это можно проделывать намного эффективнее. И тут речь не только о белках, но и о полусинтетических организмах с новыми свойствами.
Причем не стоит забывать, что, добавив всего лишь две буквы, мы расширяем число возможных генетических слов до 216, и в результате получаем возможность кодировать еще 172 аминокислоты, вдобавок к прежним двадцати. Простор для биоинженерии, как видим, бескрайний.
В десятку наиболее выдающихся результатов 2014 г. вошли работы исследователей из Скриппсовского института (Ла-Холья, США), которые не только создали ДНК с двумя новыми «буквами» генетического кода, но и заставили ее воспроизводиться внутри кишечной палочки – традиционного экспериментального объекта молекулярных биологов
Как всем известно со школьной скамьи, «текст» молекулы ДНК – хранилища наследственной информации – написан всего четырьмя «буквами»: A, T, G и C. Буквы эти обозначают четыре азотистых основания – аденин, тимин, гуанин и цитозин, которые крепятся к сахарофосфатному остову. Две такие конструкции и составляют знаменитую двойную спираль ДНК, которая в итоге содержит все инструкции по постройке и работе нашего организма.
Две цепи ДНК связаны друг с другом по принципу комплементарности: напротив A в одной цепи всегда стоит T в другой, а напротив G – всегда C. Между «противоположными» основаниями в этих парах образуются так называемые водородные связи, что и определяет соответствие оснований друг другу. Например, между A и C такие связи образоваться не могут, поэтому и говорят, что основания подходят друг к другу как ключ к замку.
Вероятность такого события очень мала, поэтому неудивительно, что если даже некогда и существовали комплементарные пары с другими основаниями, они просто не прошли через это «бутылочное горлышко» эволюции. Впрочем, первой молекулой-репликатором, скорее всего, была не ДНК, а РНК, которая, кстати сказать, вместо тимина использует еще одно азотистое основание – урацил, также образующий пары с аденином. При переходе к «миру ДНК» урацил заменился на тимин по причинам, связанным с надежностью хранения информации.
Самое интересное, что и сейчас известны организмы, у которых «великолепная четверка» отличается от описанной в школьном учебнике. Например, у многих бактериофагов – вирусов, поражающих бактерии, место тимина в ДНК занимает урацил либо гидроксиметилурацил или другие производные урацила с дополнительно присоединенным углеводным остатком. Такая «подмена» помогает вирусу защищаться от охранных систем бактерий, расщепляющих проникшую внутрь чужеродную ДНК. А в 1970-х гг. в обыкновенной луже ленинградские микробиологи обнаружили бактериофаг, у которого аденин полностью заменен другим основанием, 2,6-диаминопурином.
Что касается второго вопроса, то он лег в основу нового направления молекулярной биологии – создания искусственного генетического кода. Специалисты в этой области занимаются не только поиском возможностей создания альтернативных пар оснований, но и способов введения в структуру белков неканонических аминокислот (как известно, генетические системы всех известных на сегодня живых организмов кодируют ровно 20 «стандартных» аминокислот). Понятно, что если научиться собирать ДНК из расширенного репертуара пар оснований и заложить в код возможность включения в белки нестандартных аминокислот, то это откроет невиданные перспективы перед синтетической биологией – областью науки, занимающейся созданием несуществующих в природе живых систем и процессов.
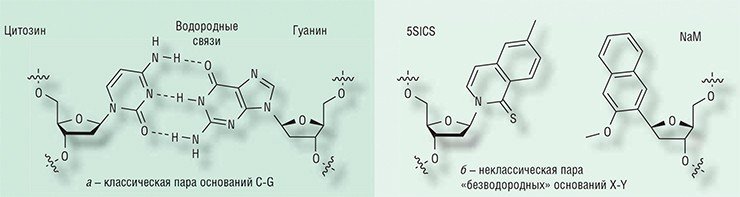
В лаборатории Ф. Роумсберга, удостоенной внимания редакторов Science, неклассическими основаниями занимаются уже не первый год. Но до недавних пор все исследования в этом направлении выполнялись in vitro, т. е. «в пробирке», а не в живой клетке. В этот раз исследователи взяли одну из таких «безводородных» пар оснований и попытались заставить ее воспроизводиться внутри бактерии кишечной палочки, традиционно используемой для экспериментов молекулярными биологами.
Однако в живом организме основания не возникают по желанию экспериментатора. За каждой из четырех букв в ДНК стоит многоходовая схема их синтеза в клетке и, разумеется, основания, придуманные химиками, клетка сама делать не может. Поэтому ученые схитрили: они ввели в бактерии белок из клеточной стенки диатомовой водоросли Phaeodactylum tricornutum, который способен захватывать те самые отдельные «буквы» ДНК непосредственно из внешней среды. Соответственно, ненатуральные основания (точнее, не сами основания, а дезоксинуклеозидтрифосфаты – «кирпичики» с частью сахарофосфатного остова, из которых и строится ДНК) просто добавляли в культуральную среду, в которой росли такие бактерии.
Но существовала еще одна проблема, которую требовалось разрешить. Дело в том, что если заставить бактериальные клетки использовать ненатуральные основания в большом количестве, то бактерии просто не выживут, потому что существующий генетический аппарат не будет их «узнавать». Поэтому ученые ввели лишь одну-единственную ненатуральную пару, и то не напрямую в саму бактериальную ДНК, а в плазмиду – отдельную маленькую кольцевую молекулу ДНК, способную существовать и самовоспроизводиться внутри бактериальной клетки. А поскольку фермент ДНК-полимераза III, которая отвечает у бактерий за репликацию основной части генома, нестандартные основания вообще не узнает, ненатуральную пару оснований пришлось ставить даже не просто в плазмиду, а в очень маленький участок плазмиды, который синтезируется другим ферментом – ДНК-полимеразой I.
После всех этих манипуляций бактерии растили на обогащенной нестандартными основаниями среде в течение 15 часов – за это время клетки успевали поделиться 24 раза. Затем определяли, что находится в том месте плазмиды, где стояла ненатуральная пара. Если бы клетка не была способна при репликации использовать соответствующие друг другу неканонические нуклеотиды, а встраивала напротив них нормальные, то ненатуральная пара после 24 делений сохранилась бы только в 1 случае из 17 млн! Однако на самом деле ничего подобного не произошло: ненатуральная пара сохранялась в 86 % случаев, замена произошла только спустя нескольких суток дальнейшего роста.
Важность работы Роумсберга и его коллег неоспорима, ведь им действительно впервые удалось показать работоспособность ненатуральной пары оснований в живой клетке. Но говорить о том, что в ней удалось «расширить генетический алфавит», очень и очень преждевременно – эта фраза вынесена в заголовок статьи в Nature явно с рекламными целями. В конце концов, авторы статьи обошли самые главные неразрешенные проблемы искусственного генетического кода. Ведь для того, чтобы на деле расширить алфавит ДНК, нужно как минимум встроить в клетку пути синтеза неканонических нуклеотидов, сделать их совместимыми с основной системой репликации и, главное, придумать, как при помощи новых букв заставить клетку производить и новые белки.
Задача по-прежнему выглядит чрезвычайно сложной – примерно, как полет в космос в эпоху начала аэронавтики. В этом смысле работу Роумсберга и его коллег можно сравнить с запуском шара братьев Монгольфье. Но в космос в конце концов полетели не воздушные шары, так что хотя отмеченное Science достижение – безусловно, шаг в нужном направлении, пока непонятно, приведет ли к цели именно эта дорога.
Литература
Власов В. В., Воробьев П. Е. Мир РНК: вчера и сегодня // НАУКА из первых рук. 2012. № 3(45). С. 40—49.
Malyshev D. A., Dhami K., Lavergne T., et al. A semi-synthetic organism with an expanded genetic alphabet // Nature. 2014. V. 509. N. 7500. P. 385—388.




